Tras las 7 actividades chulas sobre las células, los tejidos, las plantas y la fotosíntesis, ¡esta sección continúa con la fabulosa Biología Molecular!
El mundo de la PCR, la secuenciación, los plásmidos y las enzimas de restricción es apasionante y maravilloso. Especialmente si, para explicar dichos conceptos, en los centros contásemos con un equipo de electroforesis, un termociclador y 1500 cosas más.
Son materiales muy caros que, obviamente, son inexistentes en los laboratorios de Secundaria. Por tanto, como no es posible crear ratones fluorescentes con nuestro alumnado, a continuación os traigo algunas alternativas muy baratitas (y otras no tanto) para clarificar este tremendo lío del ADN recombinante.
Las opciones más asequibles son los laboratorios virtuales o los modelos recortables en papel. Aunque, si contáis con un poquito de presupuesto, otra alternativa muy chula es posible. Ya veréis…
Las 7 actividades chulas sobre Biología Molecular que os explicaré a continuación no me las he inventado yo, ni mucho menos. Algunas son traducciones/ adaptaciones de otras ideas que he encontrado por Internet o modelos DIY de kits educativos bastante caros a la venta en países anglosajones. Otras son directamente de otros autores y están disponibles desde hace tiempo en la red, pero quizá no os habíais topado todavía con ellas. Y ese es el objetivo de esta entrada: ¡encontrar estas joyitas!
Todas las actividades pueden utilizarse tanto en 4º ESO como en 1º BAC, dependiendo del nivel de la clase. Aunque algunas cosas no estén como tales en el currículo, luego aparecerán en el temario de 2ºBAC así que, el tiempo no se ha perdido… ¡Comenzamos!
1. Prueba de PATERNIDAD con sondas de papel
Si vuestro alumnado ve algo escrito en inglés y ya pone cara de pocos amigos, este es vuestro recurso. Elaborado por el IES Rosa Chacel (Colmenar Viejo, Madrid) la simulación del análisis de ADN paterno está disponible online en castellano y listo para imprimir en la página 40 (práctica nº 10) de este archivo.
La práctica te cuenta una historia rocambolesca, al más puro estilo del típico telefilm de las tardes de Antena 3, en la que una madre de alquiler (gestación subrogada tradicional /parcial) engaña a la pareja contratante y se embaraza de su propio marido. Al final, una prueba de paternidad dilucidará quién se quedará con la custodia del niño. Con estos ingredientes tan picantes, ¡les va a encantar!

Hay que recortar los fragmentos de ADN del niño, la madre de alquiler, los 2 posibles padres y el patrón, realizar una digestión (con unas tijeras a modo de enzimas de restricción) e hibridar los fragmentos obtenidos con sondas fluorescentes (también de papel, pero subrayadas con rotulador fluorescente). Después, se realizará una electroforesis por tamaños en otro folio en blanco para comprobar con qué supuesto padre coincide el patrón de bandas.
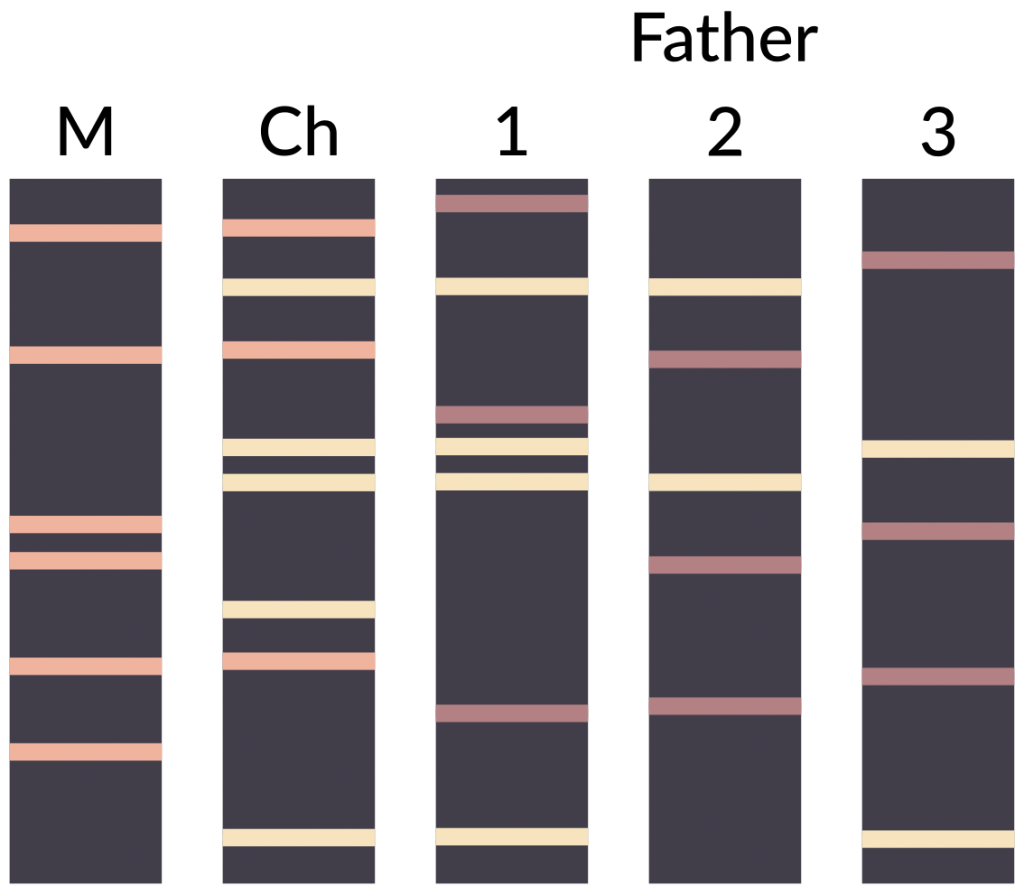
2. CSI: electroforesis para resolver un crimen
Si el culebrón de la madre de alquiler no os ha convencido, siempre podéis recurrir a Grissom y al CSI. Ha habido un robo en el museo, se han llevado las joyas de la corona. Vuestro alumnado debe transformarse en la más prestigiosa policía científica para resolverlo. Tendrán una muestra de sangre (con ADN) presente en la ventana por la que el ladrón huyó y también muestras de ADN de 4 sospechosos. Tras una digestión, al comparar el patrón de bandas en la electroforesis, el verdadero ladrón no podrá escapar de una larga sentencia condenatoria.
El archivo original de esta actividad, creado por profesores de la Towson University, en inglés, está disponible aquí o aquí (imprimid las páginas 25-28).
Aquí os dejo un pequeño video (en inglés) que va como anillo al dedo (no son las joyas de la corona sino el robo en una casa pero para el caso, nos sirve) para explicar el proceso a seguir en la actividad (extracción de ADN, digestión y posterior electroforesis):
Como veréis, esta actividad es más sencilla que la anterior, pues no utiliza sondas fluorescentes pero en la electroforesis hay que contar los pares de bases de forma cuantitativa y no solo basarse en el tamaño aproximado. La decisión de elegir una u otra dependerá del del grupo con el que la realicéis…
Si queréis realizar la actividad en castellano, podéis imprimir simplemente la página en la que aparecen las secuencias de ADN (página 27) y utilizar esta mini-presentación (en la que me he inventando cositas, la verdad) que os he colgado en Slideshare:
Además, en ambos casos, necesitaréis una copia del gel de electroforesis para pegar los fragmentos que, al no estar disponible en la red, he intentado recrear en este archivo.

* Al recortar por la diana del enzima de restricción utilizado, CC|GG, deberían quedar extremos romos. Sin embargo, como las letras de las 2 cadenas, especialmente en el sospechoso #2 y #4, no coinciden exactamente, pueden llegar a parecer extremos cohesivos. Se debe avisar al alumnado que no corten en línea recta, sino justo por el medio de las CC y las GG.
3. biotecnología: Obtención de insulina CON BACTERIAS
Para poder realizar la simulación del proceso industrial real mediante el que se obtiene la insulina que se inyectan los diabéticos, podéis descargaros el archivo aquí.
Solo necesitaréis papel normal (para imprimir los enzimas de restricción), papel de otros 2 colores distintos (para imprimir el plásmido y el gen de interés), tijeras y celo.
Será la transformación más barata del mundo…
Si queréis refrescar conceptos sobre cómo se puede insertar un gen (en este caso de la insulina humana) en un plásmido y luego transformar bacterias (a nuestra archiconocida Escherichia coli) para que produzcan cantidades industriales de insulina, podéis ver este video en el que se inserta un gen de rana en un plásmido bacteriano. De hecho, la parte que explica con el papel me parece genial para usarlo en clase, haciendo de traductor automático, of course.
Este es el protocolo a seguir en la simulación:
- El alumnado se dispone por parejas o tríos con el material citado anteriormente. No hace falta imprimir todo el archivo en inglés, solo las hojas con el fragmento del ADN genómico, el plásmido y las enzimas de restricción.

- Un alumno corta las tiras del ADN humano donde está el gen de la insulina y los pega con celo en orden una detrás de otra. Se supone que no es el genoma entero sino una amplificación por PCR de la zona del gen de la insulina.
- Mientras su pareja coloreará de colores el origen de replicación y la resistencia a kanamicina que serán elementos obligatorios en nuestro plásmido. Luego cortará las tiras del plásmido, pero esta vez tendrá que desechar dos de las tiras a elegir entre la 1 y la 6 (tirar una de las tiras sin marcadores) y desechar otra tira entre la 2 y la 3. ¡Ojo! Nunca se puede desechar la tira que tiene el origen de replicación porque si no el plásmido no se replicará y no servirá de nada. Tampoco el plásmido puede quedarse sin la resistencia a kanamicina. Asimismo, deberá tener una resistencia a antibióticos adicional y una tira sin marcadores.
 Una vez el alumnado se quede con solo 4 tiras, podrá unir los fragmentos en el orden que quiera. Además, a diferencia del ADN genómico humano, el plásmido es circular así que se deberán unir los extremos y que quede un círculo. *Si veis muy complicado que desechen esas tiras en concreto, pueden ponerlas todas y ya está, aunque el plásmido quedará excesivamente grande y difícil de manejar.
Una vez el alumnado se quede con solo 4 tiras, podrá unir los fragmentos en el orden que quiera. Además, a diferencia del ADN genómico humano, el plásmido es circular así que se deberán unir los extremos y que quede un círculo. *Si veis muy complicado que desechen esas tiras en concreto, pueden ponerlas todas y ya está, aunque el plásmido quedará excesivamente grande y difícil de manejar. - También se corta la ligasa y las distintas enzimas de restricción (todas dejan extremos cohesivos).
- Después, en equipo, deberán encontrar una enzima que corte al fragmento de ADN genómico por 2 partes pero quedando el gen de la insulina entero entre los cortes. Ese mismo enzima deberá cortar por 1 solo sitio también al plásmido.

- Una vez realizada la digestión, la ligasa unirá los enzimas cohesivos y, con suerte, el plásmido tendrá el inserto del gen de la insulina en su interior.
- Posteriormente, se puede comentar el proceso indicando cómo se introduce ese plásmido en células de E. coli (transformación) y cómo podríamos saber qué células tienen el plásmido y cuáles no (los genes de resistencia a antibióticos). Hay un laboratorio virtual más abajo que viene que ni pintado.
*Aunque la idea es genial, tiene pequeños fallos de aplicación que he intentado solventar poniendo límites para desechar 2 de las tiras (en el protocolo original solo obligan a que esté la tira con el origen de replicación). Hay varias enzimas de restricción que cortan 1 vez el plásmido y 2 veces el ADN genómico (antes y después del gen de la insulina). Estas son las posibilidades:
- HindIII (enzima de restricción producida por la bacteria Haemophilus influenzae) que corta la tira 6 del plásmido y también antes y después del gen de la insulina. Si el alumnado ha desechado la tira 6, no será una opción.
- EcoRI (producida por Escherichia coli) que aunque corta el plásmido 2 veces (en la tira 1 y entre la tira 5 y 6) en la práctica, al desechar una de esas tiras cortará el plásmido una sola vez o incluso podrá no cortarlo.
- BamHI (producida por Bacillus amyloliquefaciens) que corta el plásmido únicamente por la resistencia a la kanamicina y también corta antes y después del gen la insulina (dejando un buen trozo delante, asegurando así que no se corta ninguna región anterior importante).

El enzima de restricción idóneo es BamHI, ya que, si bien la resistencia a antibióticos nos podrá decir que bacterias se han transformado y tienen un plásmido en su interior, nunca se podrá saber si el plásmido tiene el inserto o no. Esto se debe a que la ligasa puede haber unido los extremos cohesivos del plásmido de nuevo y este haberse quedado vacío, sin inserto. Por eso, HindIII o EcoRI podrán indicarnos que bacterias tienen el plásmido pero sin asegurar que este tiene dentro el inserto. En cambio, BamHI corta la kanamicina, por lo que las bacterias transformadas con un plásmido que sí tenga el gen de la insulina, serán resistentes al otro antibiótico pero nunca a la kanamicina. Para comprobar esto, se preparan placas con ambos antibióticos por separado y se hace una réplica en placa de las colonias.
De todas formas, si os parece demasiado complicado, podéis dar por buenas todas las respuestas, siempre que corten una sola vez al plásmido y 2 veces al ADN genómico, incluyendo el gen de la insulina al completo.
4. ingeniería genética: plásmidos con «tangles»
Para crear esta actividad me he basado en el siguiente kit de Edvotek y sus instrucciones, un kit carísimo y que, para colmo, en principio no se envía a España. Súper accesible todo él.
No obstante, no todo está perdido. Puedes comprar «tangles» por un poco más de 2,5 €/cada uno aquí o aquí. Los sets que yo compré ya no están disponibles, lamentablemente. He de decir que además de «plásmidos» son una herramienta genial para desestresarse. ¡Menudo vicio!
En la actividad con los «tangles» he decidido basarme en el plásmido pUC19, aunque en vez de poner el sitio múltiple de clonaje (polylinker), he distribuido dianas de restricción para 3 enzimas distintos a lo largo del plásmido con meros fines didácticos. 
El pUC19 no tiene varias resistencias a diferentes antibióticos como el plásmido de papel anterior, sino un solo gen de resistencia a la ampicilina y el marcador LacZ, en el que aparece la diana de restricción donde se insertará el gen deseado.
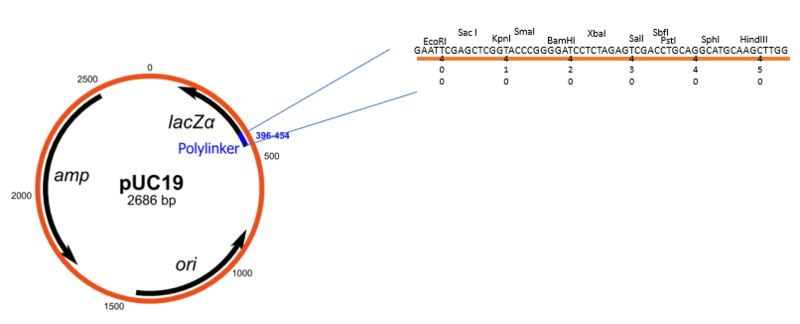
El marcador LacZ, que codifica para la enzima β-galactosidasa, evita tener que hacer las incómodas réplicas en placa del gen de resistencia a kanamicina anterior, sustituyendo ese engorroso procedimiento por una simple visualización del cambio de color de las colonias de E. coli al poner en la placa el sustrato de esta enzima, el X-Gal.
Pero, volviendo a la actividad…
A cada grupo de 4-5 personas se les repartirá un plásmido y un fragmento de gen amplificado por PCR, ambos construidos con trozos de diferentes colores de Tangles. Además, individualmente deberán ir completando esta hoja de respuestas. En realidad, se trata de una explicación magistral en la que ellos deben ir contestando una serie de actividades, pero con un twist.
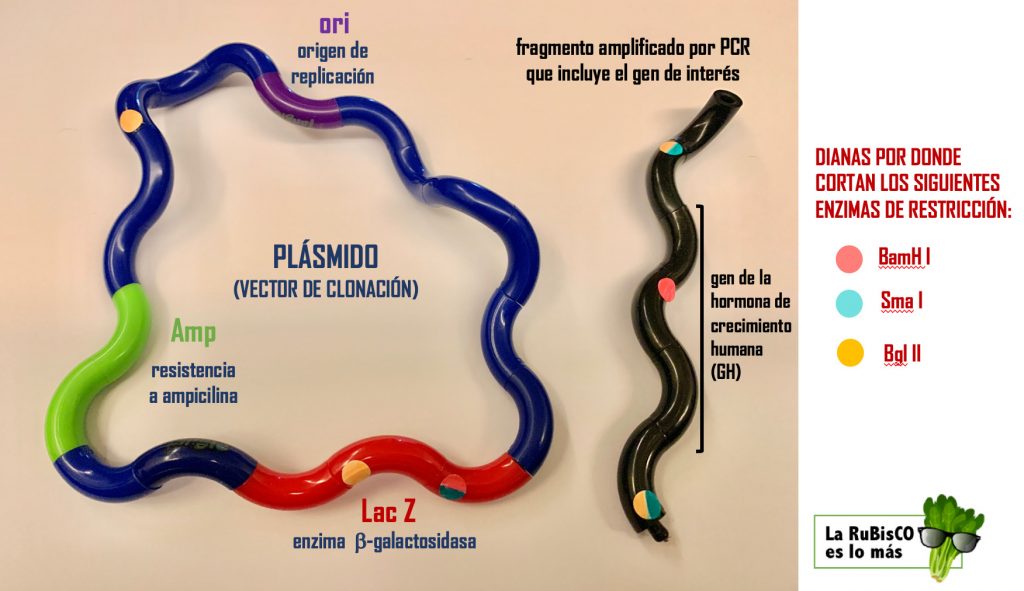
*Algunos trozos de los tangles cuestan bastante de unir, por eso, en vez de hacer que ellos construyan el plásmido (como pensé en un principio), es preferible que os arméis de paciencia y los montéis vosotros con antelación. Para marcar donde cortan los enzimas de restricción, podéis usar gomets o trocitos de etiquetas adhesivas pintadas con rotulador.
Una vez los grupos tienen la hoja de actividades y los tangles, utilizo la siguiente presentación en clase:
Si todavía dudáis si haceros o no con un set de Tangles, debo decir que, desde que los tengo en mi poder, los he utilizado un montón de veces:
- Me sirven en 1ºESO para explicar que ese «cerebro aplastado» que tienen los dibujos de las células procariotas, no es más que un círculo de ADN muy enrollado que está por ahí suelto, no protegido por el núcleo.

Se pueden usar los tangles para representar la desnaturalización de las proteínas o la estructura terciaria (superenrollamiento) del ADN circular bacteriano. - También los utilizo en 2º BAC para explicar la estructura terciaria o superenrollamiento del ADN circular de bacterias.
- En el tema de proteínas, los utilizo para representar la desnaturalización de proteínas globulares que vuelven a la estructura primaria, perdiendo su función.
- Incluso voy a usarlos en 4º ESO para explicar visualmente por qué decimos que la replicación del ADN es semiconservativa.
5. laboratorios virtuales con ADN
Por otro lado, si no os apetece que la clase se llene de papelorios recortados por el suelo ni pretendéis compraros los «tangles«, el aula de informática es otra posibilidad también muy plausible.
Existen un montón de laboratorios virtuales disponibles en la web. Una simple búsqueda en Google y encontraréis un montón. El problema es que prácticamente todos están en inglés y muchos utilizaban flash y ya no están disponibles o están reconvirtiéndose. Es lo que hay.
Respecto a la Biología Molecular, me gusta especialmente la página de «Learn Genetics» de la University of Utah. Entre los laboratorios virtuales que allí aparecen, destaca especialmente el de la preparación de la PCR que, aunque temporalmente no está disponible, lo estará otra vez en pocos meses:
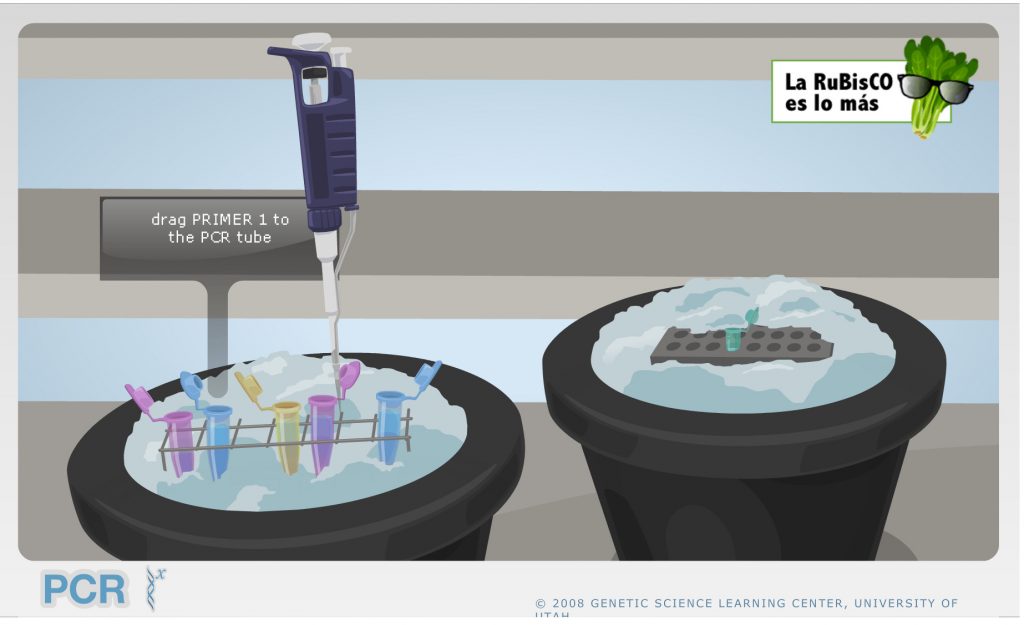
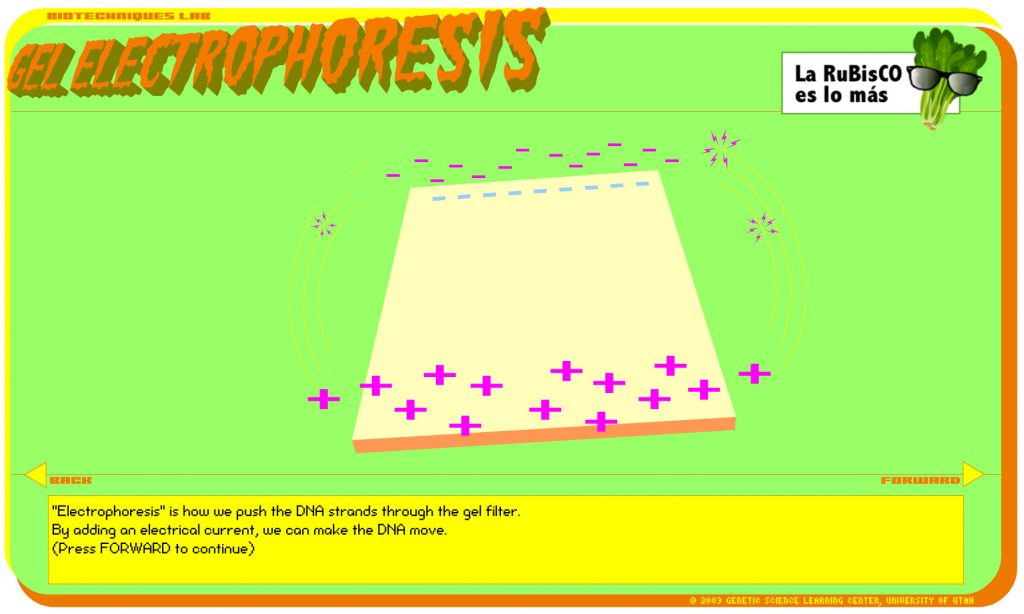
También está bien ( y todavía accesible) aunque es un poco psicodélico para mi gusto, el de la electroforesis de ADN en un gel de agarosa:
6. secuenciación y genómica: mutaciones y cáncer
Aunque las siguientes actividades entran más en el campo de las herramientas utilizadas en bioinformática, al no utilizar ordenadores y hacerlas «a mano», he decidido incluirlas en esta sección. Más adelante haré una entrada sobre algunas actividades que pueden hacerse sobre bioinformática pero, obviamente, con un ordenador y en el aula de informática.
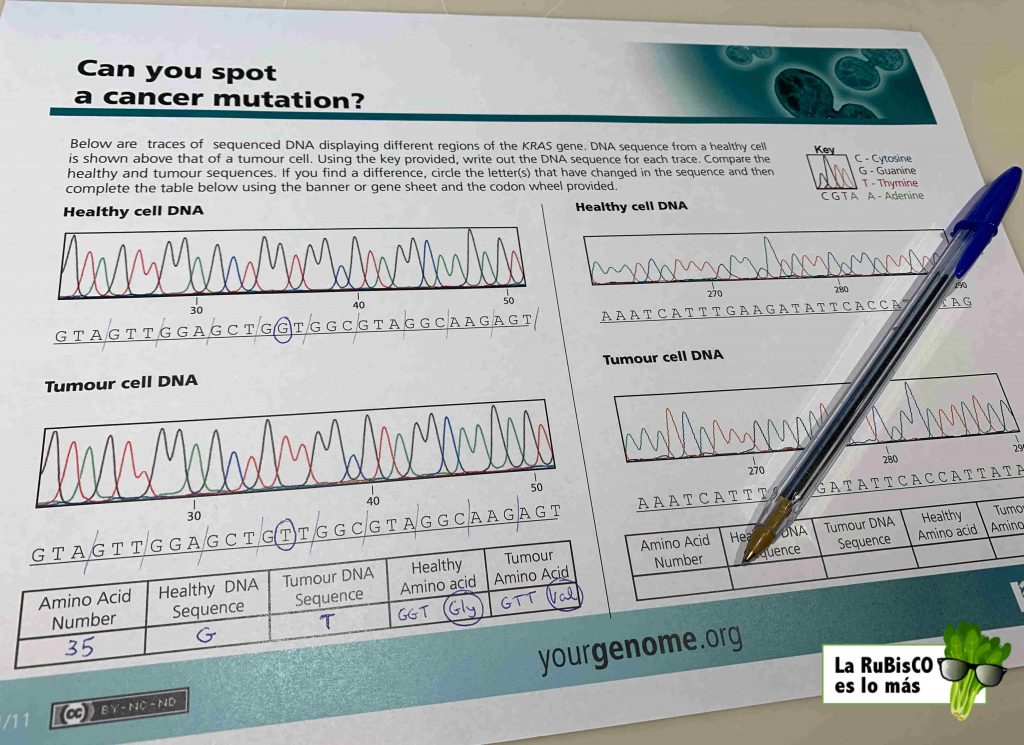
La página web YourGenome tiene un montón de actividades relacionadas con la genómica, desde protocolos de laboratorio hasta prácticas de bioinformática utilizando bases de datos biológicas reales. No obstante, yo me centraré en la actividad «KRAS: Cancer Mutation» en la que se comparan secuencias del gen KRAS, en el que la presencia de ciertas mutaciones está relacionada con el cáncer de pulmón, el de páncreas y el colorrectal.
En la página aparece tanto un PowerPoint explicativo como las hojas para entregar directamente al alumno y la ayuda para el profesor. Está genial, pero claro, todo está in English. De todas formas, si lo explicáis en clase, es muy fácil de seguir. Consiste en comparar diferentes secuencias del gen KRAS de un paciente con la secuencia KRAS de personas sanos y detectar posibles mutaciones. Es una ocasión ideal para hablar del cáncer, de la importancia del control del ciclo celular y de los oncogenes, relacionándolo así con la Biología Molecular. El único problema es que al imprimir en blanco y negro, se pierde el encanto de las gráficas de colores de las secuencias.
7. Blast sin ordenador: cladogramas «A MANO»
En esta 7ª actividad chula, como en la anterior, también habrá que comparar secuencias pero esta vez no de ADN sino de aminoácidos. Los aminoácidos aparecen representados con el código de una sola letra; no es el que utilizamos normalmente en clase, pero es el utilizado para comparar secuencias. Además, no solo tendremos la secuencia de un paciente sano y otro con cáncer sino contaremos con secuencias de varias proteínas homólogas en distintas especies animales.
Estas diferencias en los aminoácidos de una proteína altamente conservada a lo largo del tiempo en distintas especies, en este caso el citocromo C, nos ayudarán a realizar una especie de cladograma muy simple con nuestro alumnado, parecido al que se muestra en la imagen.
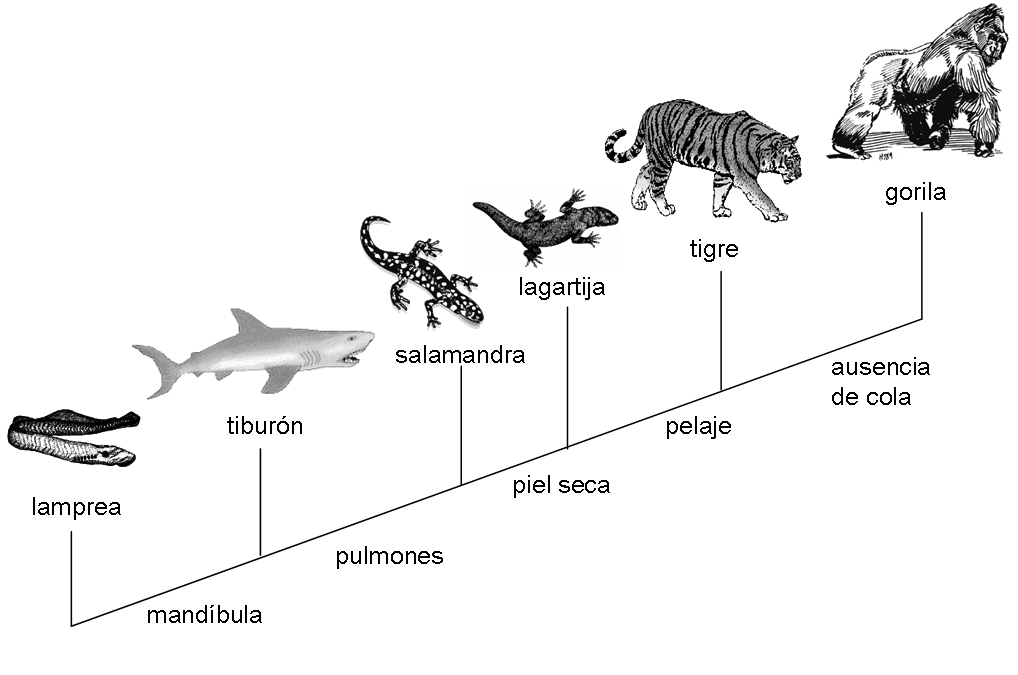
Se trata, por tanto, de una actividad genial para relacionar conceptos de Biología Molecular con el tema de la evolución de las especies.
Si queréis introducir antes a vuestro alumnado cómo realizar un cladograma, de forma simplificada, os dejo este video de Khan Academy:
Cuando vuestro alumnado comience a comparar secuencias del citocromo C en diferentes especies, se dará cuenta de lo complicado que esto resulta. De hecho, en la vida real, nadie realiza «a ojo» estas comparaciones sino que usan una herramienta informática online de alineamiento de secuencias llamada BLAST (Basic Local Alignment Search Tool). De hecho, tras la actividad, una ampliación podría ser buscar las secuencias de aminoácidos del citocromo C en dos especies y compararlas, esta vez con un ordenador mediante BLAST.

Esta actividad tampoco me la he inventado yo, sino que la modifiqué y la traduje hace ya varios años de algún lugar recóndito de Internet. El problema es que no me acuerdo de la web ni consigo encontrar la fuente original. De todas formas, aquí os dejo la actividad en Slideshare y, con mayor calidad, aquí:
Y no podría acabar este post sin mencionar la nueva revolución en la edición genética: el CRISPR-Cas9, que hace que todo lo anterior quede obsoleto. Además, CRISPR-Cas9 fue descubierto por el ilicitano Francis Mojica mientras estudiaba los microorganismos de las salinas de Santa Pola. Si queréis introducir esta técnica en clase, nada mejor que esta pegadiza canción de Julinkyvideos:
Y me despido por hoy, ¡espero que estas actividades os parezcan tan chulas como a mí!




¡Hola!
Para los que me lo habéis pedido, ya están actualizados los links y eliminados los laboratorios virtuales en Flash que, desgraciadamente, ya no funcionaban…
Buenos días, Fàtima
El post de la prueba de paternidad del IES Rosa Chacel. Al darle click al archivo no se descarga. Puede ser que ya no este? Gràcias,
Hola Estela,
A mí sí que se me descarga. No sé que ocurre, la verdad. De todas maneras, te lo envío por email.
Hola,
Qué web tan buena para los profes, nos aporta un material buenísimo para trabajar en clase de otra manera.
Muchas gracias por tu labor